DNA损伤和修复
DNA损伤和修复的机制
细胞DNA的损伤与突变和癌症的发生有关。人类细胞中的DNA每天要经历数千到数百万次的破坏事件,这些事件是由外部(外源性)和内部代谢(内源性)过程产生的。细胞基因组的改变会在DNA转录中产生错误,并随后翻译成信号传递和细胞功能所必需的蛋白质。如果突变在有丝分裂之前没有修复,基因组突变也可以传递到子代细胞。一旦细胞失去有效修复受损DNA的能力,就有三种可能的反应(图1)。
?
1.?细胞可能会衰老,即不可逆地休眠。2005年,多个实验室报告说,在体内和体外,癌细胞都可能发生衰老,阻止有丝分裂,并阻止细胞进一步进化。
2.?细胞可能发生凋亡。足够的DNA损伤可能触发凋亡信号级联,迫使细胞进入程序性细胞死亡。
3.?细胞可能变成恶性的,也就是说,发展出不朽的特征并开始不受控制的分裂。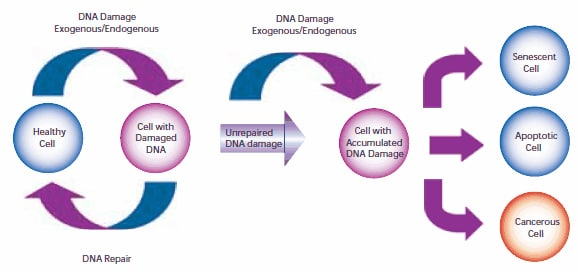
图1所示。导致衰老、细胞凋亡或癌症的细胞DNA损伤和修复的途径
为了补偿DNA损伤的程度和类型,细胞已经发展出多种修复过程,包括错配、碱基切除和核苷酸切除修复机制,几乎没有过程冗余。如果发生了压倒性的损伤,而不是消耗能量来有效地修复损伤,细胞可能已经进化到凋亡或衰老。细胞修复的速度取决于细胞类型和细胞年龄等因素。
DNA损伤的来源
多年来,外源性损伤被认为是DNA突变导致癌症的主要原因。然而,Jackson和Loeb提出,DNA损伤的内源性来源也对导致恶性肿瘤的突变起着重要作用环境和细胞来源都可能导致类似类型的DNA损伤。
?
DNA可以被物理和化学诱变剂攻击。物理诱变剂主要是辐射源,包括来自太阳的紫外线(200-300纳米波长)辐射。紫外线辐射产生共价键,交联相邻的嘧啶(胞嘧啶和胸腺嘧啶)碱基在DNA链。电离辐射(x射线)通过在细胞内产生自由基引发DNA突变,自由基产生活性氧(ROS),并导致双螺旋中的单链和双链断裂。化学诱变剂可以将烷基共价连接到DNA碱基上;能使DNA碱基甲基化或乙基化的氮芥化合物是DNA烷基化剂的例子。致癌物原是化学惰性前体,经代谢转化为高活性致癌物。这些致癌物可以与DNA反应形成DNA加合物,即附着在DNA上的化学实体。苯并[a]芘是一种多芳杂环,本身并不致癌。它在细胞色素P450酶的介导下经历了两次连续的氧化反应,产生苯并[a]芘二醇环氧化物(BPDE),这种致癌代谢物能够形成共价DNA加合物(图2)。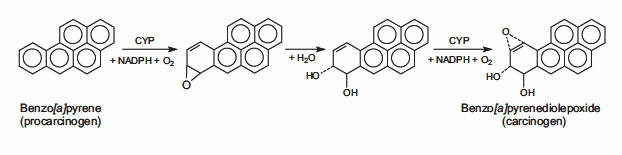
图2。苯并[a]芘被P450酶氧化生成具有高度致癌性的苯并[a]芘二烯烃。
DNA损伤也可由内源性代谢和生化反应引起,其中一些尚不清楚水解反应可以部分或完全将核苷酸碱基从DNA链上裂解。连接嘌呤碱基(腺嘌呤或鸟嘌呤)和脱氧核糖磷酸链的化学键可以在被称为脱嘌呤的过程中自发断裂。据估计,哺乳动物细胞中每天发生10,000次脱嘌呤事件脱嘧啶(从胸腺嘧啶或胞嘧啶中失去嘧啶基)也会发生,但其速率比脱嘌呤低20至100倍。
?
脱氨发生在细胞内,腺嘌呤、鸟嘌呤和胞嘧啶环的胺基丢失,分别产生次黄嘌呤、黄嘌呤和尿嘧啶。DNA修复酶能够识别和纠正这些不自然的碱基。然而,在随后的DNA复制过程中,未经纠正的尿嘧啶碱基可能被误读为胸腺嘧啶,并产生C→T点突变。
?
DNA甲基化,一种特殊形式的烷基化,发生在细胞内,由于与s -腺苷蛋氨酸(SAM)的反应。SAM是一种含有高活性甲基的细胞内代谢中间体。在哺乳动物细胞中,甲基化发生在距离鸟苷基(G)?5′的胞嘧啶碱基(C)胞嘧啶环的5位,即序列CpG。突变误差的一个重要来源是甲基化的5-甲基胞嘧啶产物的自发脱氨。胺基的损失导致胸腺嘧啶碱基,它不被DNA修复酶检测到作为一个非自然的碱基。由此产生的取代保留在DNA复制中,产生C→T点突变(图3)。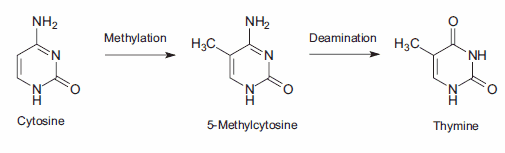
正常的代谢过程产生活性氧(ROS),通过氧化修饰碱基。嘌呤和嘧啶碱都容易氧化。最常见的突变是鸟嘌呤氧化为8-oxo-7,8-二氢鸟嘌呤,导致核苷酸8-oxo-脱氧鸟嘌呤sin (8-oxo-dG)。8-oxo-dG能够与脱氧腺苷碱基配对,而不是像预期的那样与脱氧胞苷碱基配对。如果这个错误没有被错配修复酶检测到并纠正,随后复制的DNA将包含C→a点突变。ROS还可能导致脱氧核糖核酸的脱嘌呤、脱嘧啶和单链或双链断裂。
?
在细胞周期S期的DNA复制过程中可能引入其他基因组突变。复制模板DNA的聚合酶有一个小但显著的错误率,并且可能包含基于沃森-克里克配对的与模板DNA不正确的核苷酸。化学改变的核苷酸前体可以被聚合酶合并到生成的DNA中,而不是正常的碱基。此外,当复制含有大量重复核苷酸或重复序列的DNA片段(微卫星区)时,聚合酶容易“口吃”。这种酶的“口吃”是由于链滑移,当模板和复制的DNA链脱离正确的排列时。结果,聚合酶无法插入模板DNA所指示的正确数量的核苷酸,导致子链中的核苷酸过少或过多。
?
DNA可能发生单链和双链裂解。单链断裂可能是由于脱氧核糖基磷酸链的脱氧核糖部分受损造成的。断裂也是ap -核酸内切酶去除磷酸脱氧核糖后碱基切除修复途径的中间步骤。当单链断裂发生时,核苷酸碱基和脱氧核糖主干都从DNA结构中丢失。双链裂解最常发生在细胞经过s期时,因为DNA在展开作为复制模板时更容易断裂。
?
DNA修复机制
虽然细胞能够进化到凋亡或衰老状态,但这些行动是最后的手段。对于每一种类型的DNA损伤,细胞已经进化出一种修复损伤或消除破坏性化合物的特定方法。
?
o6 -甲基鸟嘌呤DNA甲基转移酶(MGMT;DNA烷基转移酶)根据DNA结构从鸟嘌呤中分离甲基和乙基加合物。该反应不是催化(酶促)反应,而是化学计量(化学)反应,每去除一个加合物就消耗一个MGMT分子。经过过表达MGMT的细胞对癌症的抵抗力更强,可能是因为它们能够抵消大量的烷基化损伤。Niture等人最近的一项研究报告称,使用半胱氨酸/谷胱甘肽增强药物和天然抗氧化剂可增加MGMT的表达。
?
DNA聚合酶,如聚合酶-δ,含有校对活性,主要参与复制错误修复。当检测到错误时,这些聚合酶就会停止DNA复制的过程,然后反向从子DNA链中移除核苷酸,直到不正确的核苷酸明显消失,然后重新启动正向复制过程。在Pold1基因的两个拷贝中都有点突变的小鼠表现出DNA聚合酶-δ的校对活性的丧失,并且与具有野生型基因或单拷贝突变的小鼠相比,发生上皮性癌症的几率显著更高。
?
一组被称为错配切除修复(MMR)酶的蛋白质能够纠正DNA聚合酶校对活动未检测到的复制错误。MMR酶从子DNA中去除一个不正确的核苷酸,并使用W-C配对和父DNA链作为正确的模板来修复链这对于微卫星区域复制过程中产生的错误尤其重要,因为DNA聚合酶的校对活动不会检测到这些错误。在较小程度上,MMR酶还纠正了由DNA氧化或烷基化引起的各种碱基对异常。这些突变包括含有o6 -甲基鸟嘌呤和8-氧鸟嘌呤的修饰碱基对,以及致癌物和顺铂加合物。12,13人错配切除修复基因MSH2和MLH1突变与遗传性非息肉病性结直肠癌(HNPCC)综合征相关。
?
碱基切除修复和核苷酸切除修复
碱基切除修复(BER)涉及多个酶切除和替换单个受损的核苷酸碱基。主要由BER酶修复的碱基修饰是内源性氧化和水解破坏的碱基修饰。DNA糖基化酶切断核苷酸碱基和核糖之间的键,使DNA的核糖磷酸链完整,但产生无嘌呤或无嘧啶(AP)位点。8-Oxoguanine DNA糖基化酶I (Ogg1)去除7,8-二氢- 8-Oxoguanine (8-oxoG),这是活性氧产生的碱基突变之一。人类OGG1基因的多态性与肺癌和前列腺癌等各种癌症的风险相关。尿嘧啶DNA糖基化酶,另一种BER酶,切除胞嘧啶脱氨的产物尿嘧啶,从而防止随后的C→T点突变n -甲基嘌呤DNA糖基化酶(MPG)能够去除多种修饰的嘌呤碱基。
?
DNA中由BER酶作用产生的AP位点,以及由脱嘧啶和脱嘌呤作用产生的AP位点,可通过AP-核酸内切酶1 (APE1)的作用进行修复。APE1将磷酸二酯链5′裂解到AP位点。DNA链含有一个3 ' -羟基和一个5 ' -碱性脱氧核糖磷酸。DNA聚合酶β (Polβ)根据相应的W-C配对插入正确的核苷酸,并通过其相关的ap裂解酶活性去除脱氧核糖磷酸。x射线修复交叉互补基团1 (XRCC1)的存在是与DNA连接酶III (LIG3)形成异二聚体所必需的。XRCC1作为一个支架蛋白,为Polβ提供一个非活性的结合位点,并在修复位点将Polβ和LIG3酶结合在一起Poly(adp -核糖)聚合酶(PARP-1)与XRCC1和Polβ相互作用,是BER途径的必要组成部分。18,19修复的最后一步由LIG3完成,它将替换核苷酸的脱氧核糖连接到脱氧核糖基磷酸主干。这一途径被命名为“短补丁误码率”。
?
另一种被称为“长补丁误码率”的途径用最少2个核苷酸的长度替换一条核苷酸链。据报道,修复长度为10到12个核苷酸。21,22 Longpatch BER需要增殖细胞核抗原(PCNA)的存在,它作为重组酶的支架蛋白其他DNA聚合酶,可能是Polδ和Polε, 24用于产生寡核苷酸瓣。现有的核苷酸序列被皮瓣内切酶-1 (FEN1)去除。然后寡核苷酸被DNA连接酶I (LIG1)连接到DNA上,封闭断裂并完成修复用于确定短补丁与长补丁误码率路径选择的过程仍在研究中(图4)。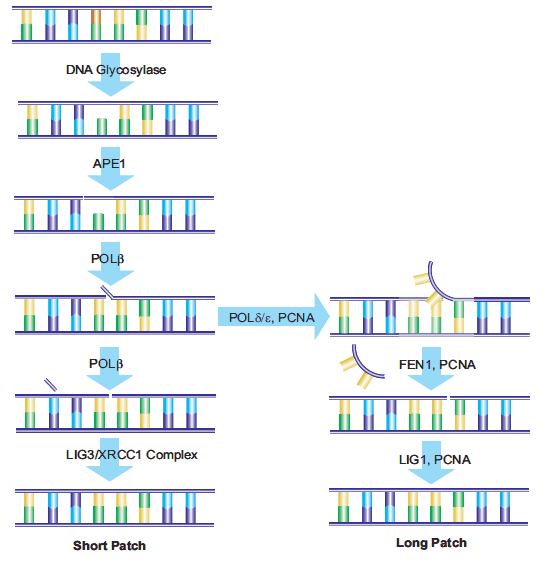
图4。短补丁和长补丁误码率路径示意图。
虽然误码率可能通过长补丁途径取代多个核苷酸,但短补丁和长补丁误码率的起始事件都是对单个核苷酸的损伤,从而对DNA双螺旋结构的影响最小。核苷酸切除修复(NER)修复至少含有2个碱基的核苷酸链的损伤,并造成DNA的结构扭曲。NER的作用是修复单链断裂以及外源的一系列损伤,如笨重的DNA加合物和紫外线辐射。同样的途径也可以用来修复氧化应激造成的损伤在哺乳动物细胞中,有超过20种蛋白质参与NER通路,包括XPA、XPC-hHR23B、复制蛋白A (RPA)、转录因子TFIIH、XPB和XPD DNA解旋酶、ERCC1- xpf和XPG、Polδ、Polε、PCNA和复制因子c。切除修复交叉互补(ERCC1)基因的过表达与非小细胞肺癌细胞对顺铂的耐药有关29,并对应于增强的DNA修复能力30全球基因组NER (GGR)修复整个基因组的损伤,而一种称为转录偶联修复(TCR)的特定NER通路在活性RNA聚合酶转录期间修复基因。
双股断裂的修复
DNA中的双链断裂会导致基因组序列的丢失和重排。这些断裂可以通过非同源端连接(NHEJ)或同源重组(HR)进行修复,也称为重组修复或模板辅助修复。
?
当细胞处于S/G2晚期且模板最近被复制时,HR通路被激活。这种机制需要存在一个相同或几乎相同的序列,通过着丝粒连接到受损的DNA区域,作为修复模板。由该机制修复的双链断裂通常是由复制机制试图通过单链断裂或未修复的病变进行合成引起的,导致复制叉的崩溃。
?
当姐妹染色单体不能用作HR模板时,非同源末端连接(NHEJ)用于细胞周期的其他点。当这些断裂发生时,细胞还没有复制包含断裂的DNA区域,因此与HR途径不同,没有相应的模板链可用。在NHEJ中,Ku异二聚体蛋白在没有可用模板的情况下定位断裂DNA链的两端进行修复,在此过程中丢失序列信息。多个酶参与了再连接过程,包括DNA连接酶IV, XRCC4和DNA依赖蛋白激酶(DNA- pk)。32,33 NHEJ具有固有的诱变性,因为它依赖于两个待连接DNA片段的单链尾部之间的偶然配对,称为微同源性(图5)。在高等真核生物中,NHEJ修复需要DNA- pk,无论是通过主要机制还是通过替代备份机制(D-NHEJ)。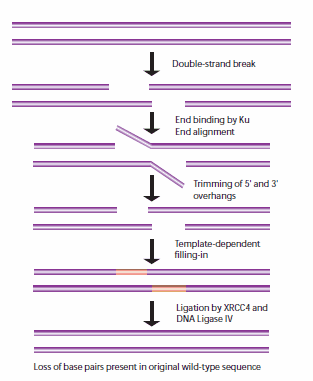
图5。NHEJ修复DNA双链断裂的一般机制。
未来的应用
虽然DNA损伤是癌细胞发育和进化的关键因素,但持续的损伤被用作癌症临床治疗的一部分,迫使恶性细胞凋亡或衰老。许多化疗药物,如博莱霉素、丝裂霉素和顺铂,都是有效的,因为它们会进一步破坏癌细胞的DNA,而癌细胞的复制速度比周围组织更快。细胞DNA修复机制是一把双刃剑;通过减少可能导致癌症的突变,这些过程努力保持基因组的完整性,但在恶性细胞中,同样的机制允许这些细胞在额外的DNA损伤中幸存下来,并继续不受控制地生长。为了阻断癌细胞内的这种生存机制,目前正在进行临床试验,使用抑制剂抑制特定的DNA修复酶,包括MGMT、PARP和DNA- pk。35-38
参考文献
1.Collado?M,?Gil?J,?Efeyan?A,?Guerra?C,?Schuhmacher?AJ,?Barradas?M,?Benguría?A,?Zaballos?A,?Flores?JM,?Barbacid?M,?et al.?2005.?Senescence in premalignant?tumours.?Nature.?436(7051):642-642.?https://doi.org/10.1038/436642a
2.Chen?Z,?Trotman?LC,?Shaffer?D,?Lin?H,?Dotan?ZA,?Niki?M,?Koutcher?JA,?Scher?HI,?Ludwig?T,?Gerald?W,?et al.?2005.?Crucial role of p53-dependent cellular senescence in suppression of Pten-deficient?tumorigenesis.?Nature.?436(7051):725-730.?https://doi.org/10.1038/nature03918
3.Michaloglou?C,?Vredeveld?LCW,?Soengas?MS,?Denoyelle?C,?Kuilman?T,?van der Horst?CMAM,?Majoor?DM,?Shay?JW,?Mooi?WJ,?Peeper?DS.?2005.?BRAFE600-associated senescence-like cell cycle arrest of human naevi.?Nature.?436(7051):720-724.?https://doi.org/10.1038/nature03890
4.Braig?M,?Lee?S,?Loddenkemper?C,?Rudolph?C,?Peters?AH,?Schlegelberger?B,?Stein?H,?D?rken?B,?Jenuwein?T,?Schmitt?CA.?2005.?Oncogene-induced senescence as an initial barrier in lymphoma development.?Nature.?436(7051):660-665.?https://doi.org/10.1038/nature03841
5.Jackson?AL,?Loeb?LA.?2001.?The contribution of endogenous sources of DNA damage to the multiple mutations in cancer.?Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis.?477(1-2):7-21.?https://doi.org/10.1016/s0027-5107(01)00091-4
6.De Bont?R.?2004.?Endogenous DNA damage in humans: a review of quantitative data.?Mutagenesis.?19(3):169-185.?https://doi.org/10.1093/mutage/geh025
7.Lindahl?T,?Nyberg?B.?1972.?Rate of depurination of native deoxyribonucleic acid.?Biochemistry.?11(19):3610-3618.?https://doi.org/10.1021/bi00769a018
8.Brem?R.?2005.?XRCC1 is required for DNA single-strand break repair in human cells.?Nucleic Acids Research.?33(8):2512-2520.?https://doi.org/10.1093/nar/gki543
9.Niture?SK,?Velu?CS,?Smith?QR,?Bhat?G,?Srivenugopal?KS.?Increased expression of the MGMT repair protein mediated by cysteine prodrugs and chemopreventative natural products in human lymphocytes and tumor cell lines.?Carcinogenesis.?28(2):378-389.?https://doi.org/10.1093/carcin/bgl155
10.Goldsby?RE,?Hays?LE,?Chen?X,?Olmsted?EA,?Slayton?WB,?Spangrude?GJ,?Preston?BD.?2002.?Nonlinear partial differential equations and applications: High incidence of epithelial cancers in mice deficient for DNA polymerase ? proofreading.?Proceedings of the National Academy of Sciences.?99(24):15560-15565.?https://doi.org/10.1073/pnas.232340999
11.Yang?W.?2000.?Structure and function of mismatch repair proteins.?Mutation Research/DNA Repair.?460(3-4):245-256.?https://doi.org/10.1016/s0921-8777(00)00030-6
12.Iyer?RR,?Pluciennik?A,?Burdett?V,?Modrich?PL.?2006.?DNA Mismatch Repair:? Functions and Mechanisms.?Chem. Rev..?106(2):302-323.?https://doi.org/10.1021/cr0404794
13.Modrich?P.?2006.?Mechanisms in Eukaryotic Mismatch Repair.?J. Biol. Chem..?281(41):30305-30309.?https://doi.org/10.1074/jbc.r600022200
14.Müller?A,?Fishel?R.?2002.?Mismatch Repair and the Hereditary Non-polyposis Colorectal Cancer Syndrome (HNPCC).?Cancer Investigation.?20(1):102-109.?https://doi.org/10.1081/cnv-120000371
15.Lindahl?T.?1974.?An N-Glycosidase from Escherichia coli That Releases Free Uracil from DNA Containing Deaminated Cytosine Residues.?Proceedings of the National Academy of Sciences.?71(9):3649-3653.?https://doi.org/10.1073/pnas.71.9.3649
16.Singer?B,?Hang?B.?1997.?What Structural Features Determine Repair Enzyme Specificity and Mechanism in Chemically Modified DNA??.?Chem. Res. Toxicol..?10(7):713-732.?https://doi.org/10.1021/tx970011e
17.Lindahl?T.?1999.?Quality Control by DNA Repair.?286(5446):1897-1905.?https://doi.org/10.1126/science.286.5446.1897
18.Caldecott?KW,?Aoufouchi?S,?Johnson?P,?Shall?S.?1996.?XRCC1 Polypeptide Interacts with DNA Polymerase ? and Possibly Poly (ADP-Ribose) Polymerase, and DNA Ligase III Is a Novel Molecular 'Nick-Sensor' In Vitro.?Nucleic Acids Research.?24(22):4387-4394.?https://doi.org/10.1093/nar/24.22.4387
19.Dantzer?F,?de la Rubia?G,?Ménissier-de Murcia?J,?Hostomsky?Z,?de Murcia?G,?Schreiber?V.?2000.?Base Excision Repair Is Impaired in Mammalian Cells Lacking Poly(ADP-ribose) Polymerase-1?.?Biochemistry.?39(25):7559-7569.?https://doi.org/10.1021/bi0003442
20.Srivastava?DK,?Vande Berg?BJ,?Prasad?R,?Molina?JT,?Beard?WA,?Tomkinson?AE,?Wilson?SH.?1998.?Mammalian Abasic Site Base Excision Repair.?J. Biol. Chem..?273(33):21203-21209.?https://doi.org/10.1074/jbc.273.33.21203
21.Ranalli?TA,?Tom?S,?Bambara?RA.?2002.?AP Endonuclease 1 Coordinates Flap Endonuclease 1 and DNA Ligase I Activity in Long Patch Base Excision Repair.?J. Biol. Chem..?277(44):41715-41724.?https://doi.org/10.1074/jbc.m207207200
22.Sattler?U,?Frit?P,?Salles?B,?Calsou?P.?2003.?Long?patch DNA repair synthesis during base excision repair in mammalian cells.?EMBO Rep.?4(4):363-367.?https://doi.org/10.1038/sj.embor.embor796
23.Fortini?P,?Pascucci?B,?Parlanti?E,?Sobol?RW,?Wilson?SH,?Dogliotti?E.?1998.?Different DNA Polymerases Are Involved in the Short- and Long-Patch Base Excision Repair in Mammalian Cells?.?Biochemistry.?37(11):3575-3580.?https://doi.org/10.1021/bi972999h
24.Klungland?A.?1997.?Second pathway for completion of human DNA base excision-repair: reconstitution with purified proteins and requirement for DNase IV (FEN1).?16(11):3341-3348.?https://doi.org/10.1093/emboj/16.11.3341
25.Sung?J,?Demple?B.?2006.?Roles of base excision repair subpathways in correcting oxidized abasic sites in DNA.?FEBS Journal.?273(8):1620-1629.?https://doi.org/10.1111/j.1742-4658.2006.05192.x
26.Balajee?AS,?Bohr?VA.?2000.?Genomic heterogeneity of nucleotide excision repair.?Gene.?250(1-2):15-30.?https://doi.org/10.1016/s0378-1119(00)00172-4
27.Gros?L,?Saparbaev?MK,?Laval?J.?2002.?Enzymology of the repair of free radicals-induced DNA damage.?Oncogene.?21(58):8905-8925.?https://doi.org/10.1038/sj.onc.1206005
28.You?J,?Wang?M,?Lee?S.?2003.?Biochemical Analysis of the Damage Recognition Process in Nucleotide Excision Repair.?J. Biol. Chem..?278(9):7476-7485.?https://doi.org/10.1074/jbc.m210603200
29.Rosell?R,?Taron?M,?Barnadas?A,?Scagliotti?G,?Sarries?C,?Roig?B.?2003.?Nucleotide Excision Repair Pathways Involved in Cisplatin Resistance in Non-Small-Cell Lung Cancer.?Cancer Control.?10(4):297-305.?https://doi.org/10.1177/107327480301000404
30.Vogel?U,?Dybdahl?M,?Frentz?G,?Nex??BA.?2000.?DNA repair capacity: inconsistency between effect of over-expression of five NER genes and the correlation to mRNA levels in primary lymphocytes.?Mutation Research/DNA Repair.?461(3):197-210.?https://doi.org/10.1016/s0921-8777(00)00051-3
31.Hanawalt?PC.?2002.?Subpathways of nucleotide excision repair and their regulation.?Oncogene.?21(58):8949-8956.?https://doi.org/10.1038/sj.onc.1206096
32.Critchlow?SE,?Jackson?SP.?1998.?DNA end-joining: from yeast to man.?Trends in Biochemical Sciences.?23(10):394-398.?https://doi.org/10.1016/s0968-0004(98)01284-5
33.Wang?H.?2003.?Biochemical evidence for Ku-independent backup pathways of NHEJ.?Nucleic Acids Research.?31(18):5377-5388.?https://doi.org/10.1093/nar/gkg728
34.Perrault?R,?Wang?H,?Wang?M,?Rosidi?B,?Iliakis?G.?2004.?Backup pathways of NHEJ are suppressed by DNA-PK.?J. Cell. Biochem..?92(4):781-794.?https://doi.org/10.1002/jcb.20104
35.Sánchez-Pérez?I.?2006.?DNA repair inhibitors in cancer treatment.?Clin Transl Oncol.?8(9):642-646.?https://doi.org/10.1007/s12094-006-0034-8
36.Madhusudan?S,?Hickson?ID.?2005.?DNA repair inhibition: a selective tumour targeting strategy.?Trends in Molecular Medicine.?11(11):503-511.?https://doi.org/10.1016/j.molmed.2005.09.004
37.PLUMMER?E.?2006.?Inhibition of poly(ADP-ribose) polymerase in cancer.?Current Opinion in Pharmacology.?6(4):364-368.?https://doi.org/10.1016/j.coph.2006.02.004
38.SABHARWAL?A,?MIDDLETON?M.?2006.?Exploiting the role of O6-methylguanine-DNA-methyltransferase (MGMT) in cancer therapy.?Current Opinion in Pharmacology.?6(4):355-363.?https://doi.org/10.1016/j.coph.2006.03.011
 首页
首页 400-620-6333
400-620-6333




 危险品化学品经营许可证(带存储)
危险品化学品经营许可证(带存储)